|
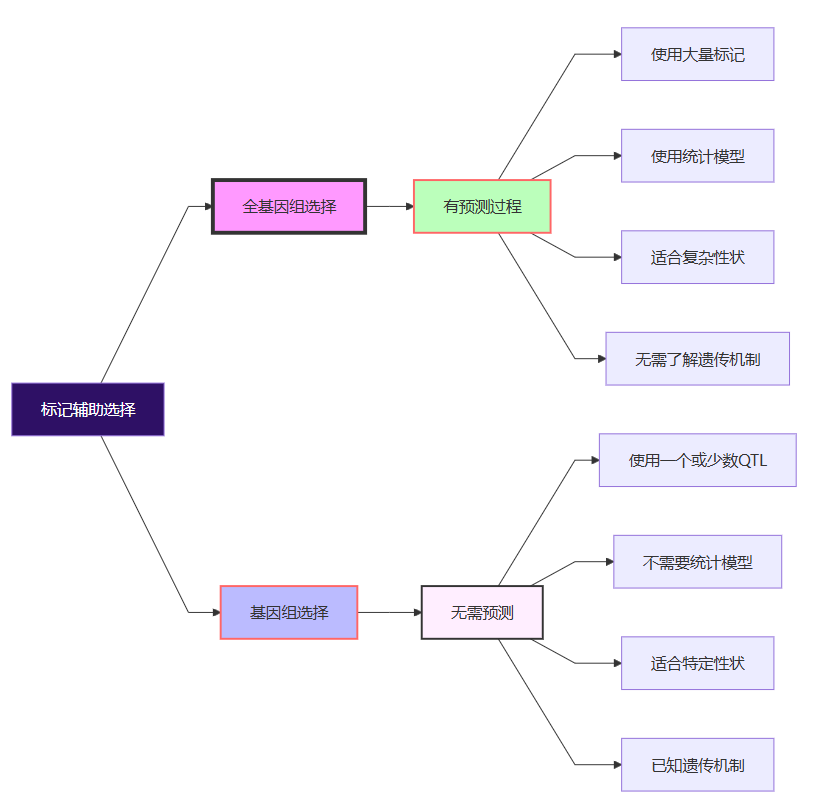
清晰的基本概念,可以更好的理解事物并实现创新。 很多人常常把Genomic selection或Genowide selection翻译成全基因组选择,而有一些人把它翻译成基因组选择(很多翻译软件或AI是这么翻译的,并被直接拿来使用)。那么,全基因组选择和基因组选择有什么区别呢? 首先,最早做GS育种的科学家比较倾向于使用全基因组选择这个概念,那么为什么要加一个“全”字呢?这来自于全基因组选择早期的概念——GS可以使用遍布整个基因组的全部分子标记来估计个体的育种值,而不是用少数几个标记。使用一个或少量分子标记/QTL进行育种材料选择的方案通常叫做分子标记辅助选择(Molecular marker assisted selection,MAS),也可以叫做基因组选择,因为它在筛选材料时,的确用到了基因组信息,但这种选择并不包含预测部分,且必须先对特定性状的遗传机制有清晰的认识,然后根据特殊表型对应的标记直接选择适合的材料。全基因组选择则不同,该方案必须经过建模和预测的过程。 2001年,全基因组选择的概念被提出时,为了强调GS是分子标记辅助选择但又区别于已知的MAS,把原有的MAS叫做传统的分子标记辅助选择,也就是基因组选择。同时,有学者把GS这套通工具,将多个学科与信息学和机器学习等新技术相结合来研究整个基因组(大量标记),以改进植物育种项目中的选择和交配,归类为基因组辅助育种(Genomic assisted breeding,GAB)。GAB分类下的另外两个工具分别是遗传转化和基因编辑。 因此,全基因组选择和基因组选择是两个不同的概念,但他们常常被混淆。有时,英文直译并不符合中文的使用标准。 全基因组选择和基因组选择的差异,请看图。  如果觉得思维导图太复杂,也可以查看全基因组选择和基因组选择的特征表。
| 特征 | 全基因组选择 | 基因组选择 | | 标记密度 | 密集 (几十万到几百万个 SNP) | 稀疏 (几百到几千个标记) | | 必须预测 | 是 | 否 | | 预测模型 | 统计模型 (RR-BLUP, BayesA等) | 无 | | 已知条件 | 亲缘关系、系谱或无 | 遗传机制 | | 应用范围 | 复杂性状 | 简单性状 | | 成本 | 高 | 低 |
附录mermaid code
- graph LR
- A[标记辅助选择] --> B[全基因组选择]
- A --> C[基因组选择]
- B --> D[有预测过程]
- C --> E[无需预测]
- D --> F[使用大量标记]
- D --> G[使用统计模型]
- D --> H[适合复杂性状]
- D --> I[无需了解遗传机制]
- E --> J[使用一个或少数QTL]
- E --> K[不需要统计模型]
- E --> L[适合特定性状]
- E --> M[已知遗传机制]
-
- style A fill:#2e1065,color:white
- style B fill:#f9f,stroke:#333,stroke-width:4px
- style C fill:#bbf,stroke:#f66,stroke-width:2px
- style D fill:#bfb,stroke:#f66,stroke-width:2px
- style E fill:#fef,stroke:#333,stroke-width:2px
|